2. 北京林业大学林木育种国家工程实验室, 林木花卉遗传育种教育部重点实验室, 北京, 100083
*同等贡献作者
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2012 年, 第 10 卷, 第 79 篇 doi: 10.5376/mpb.cn.2012.10.0079
收稿日期: 2012年11月23日 接受日期: 2012年12月07日 发表日期: 2012年12月27日
引用格式(中文):
张瑞丽等, 2012, 云南松种质资源SSR标记的引物筛选及Hardy-Weinberg检测, 分子植物育种(online) Vol.10 No.79 pp.1577-1582 (doi:10.5376/mpb. cn.2012.10.0079)
引用格式(英文):
Zhang et al., 2012, Primer Screening of SSR Markers for Germplasm of Pinus yunnanensis and applying to Hardy-Weinberg Detection, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding) Vol.10 No.79 pp.1577-1582 (doi: 10.5376/mpb.cn.2012.10.0079)
本研究以5个不同分布区域的云南松群体DNA为模板,从59对引物中筛选出重复性好、条带清晰的多态性引物7对。用筛选出的7对引物对云南松5个天然群体进行PCR扩增,共扩增出55条谱条,平均每个引物扩增出7.9谱条。所筛选出的7对引物用于云南松种质资源的Hardy-Weinberg检测。
云南松(Pinus yunnanensis Franch)是松科(Pinaceae)松属(Pinus)植物,在云南的亚热带高原,海拔700~3200 m都有大面积分布(吴征镒等, 1987, 科学出版社, pp.417-419.),是分布区域内瘠薄荒山造林的先锋树种和治理水土流失的重要树种,占云南省林地面积的52%、占有林地蓄积的32%,在分布区域林业经济生产和生态建设中占有举足轻重的作用(中国科学院中国植物志编辑委员会, 1978, 科学出版社, pp.211-282; 金振洲和彭鉴, 2004, 云南科技出版社, pp.6, 21, 332)。然而,目前云南松的退化现象比较明显,急需对现有的种质资源进行评价,以制定切实可行的遗传保护策略。
随着分子生物学的发展,人们更重视基因本身即DNA分子水平的评价。目前,在林木种质资源研究中使用比较多的有RFLP、RAPD、AFLP、SSR、ISSR等,其中,微卫星(simple sequence repeat, SSR)是以少数几个核苷酸为单位多次串联重复的DNA序列,具有数量丰富、共显性、高度重复性、对DNA数量及质量要求不高等优点(Powell et al, 1996; Kalia, 2011; Krapal et al, 2012)而得到广泛应用,如Soto等(2010)基于SSR标记分析气候变化对伊比利亚半岛当地几种松的遗传结构的影响;Eckert等(2010)利用SSR标记研究表明不同的林分类型空间遗传结构也不同。然而,为实现SSR高效扩增,除稳定的扩增反应条件外,适宜的引物是实现有效或多态扩增的关键,而引物的来源主要是利用已知序列根据其两端较为保守的序列设计(Krutovsky et al., 2004; 陈怀琼等, 2009)或利用种间可转移性从近缘种中筛选(尤卫艳等, 2010),相比而言,后者是较快捷的途径。
本研究以云南松实生苗基因组DNA为模板,对来源于松属近缘种的SSR引物进行有效扩增和多态性扩增筛选,对获得的多态性扩增引物,再用5个群体120个个体进行Hardy-Weinberg检测和多样性扩增分析。
1结果分析
1.1引物退火温度对扩增产物的影响
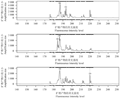
SSR-PCR扩增时的退火温度对扩增谱带式样影响明显(张冬梅等, 2007),常用一般程序和梯度降温程序进行扩增以确定退火温度,在本研究中所采用梯度降温程序(94 ℃ 4 min, 94 ℃ 30 s, T<Tm值±5 ℃> 40 s, 72 ℃ 45 s, 35循环, 72 ℃ 10 min, 4 ℃保存)确定各引物的退火温度。
由图1所示,2对引物各设置6个退火温度,随着温度的逐渐提高(图中从左至右),呈现出不同的扩增效果,温度低,非特异性扩增较明显,随着温度的增加,特异性增强,但超过一定温度范围则无扩增产物。
|
|
1.2多态性引物的筛选
1.2.1 多态性引物的选择
引物能否有效扩增主要根据条带的有无及清晰度来判断。用单一样本对59对SSR引物进行初筛,其中有23对引物能够在样品中扩增出片段,再用5个云南松群体各4个个体样品DNA进行多态性检测(荧光检测),筛选出7对多态性引物。部分引物的扩增结果如下图所示。
由图2可以看出,所筛选出来的引物在云南松群体中扩增效果较好。
|
|
1.2.2 多态性引物的Hardy-Weinberg检测
对所筛选的7个多态性引物,采用5个云南松群体120个个体进行Hardy-Weinberg检测,结果见表1。
|
|
Hardy-Weinberg杂交过剩假设检验结果表明(表1),PR118、RPTest11、PtTX4001在所有测试群体中均处于Hardy-Weinberg平衡,PtTX3091和PtTX3011在所所有测试群体中未处于Hardy-Weinberg平衡;PTest1在FN和HQ群体、PtTX3122在HQ和LX群体中存在Hardy-Weinberg平衡,而在其它几个群体存在Hardy-Weinberg不平衡。同时对连锁不平衡的检测可知,各位点间无明显的连锁不平衡,完全可用于云南松SSR分析。
1.2.3 多态性引物的多态性扩增参数分析
用5个云南松群体120个个体,对所筛选的7个多态性引物进行多态性扩增参数分析,结果见表2。
|
|
从表2可以看出,7个位点在5个供试群体中共检测出的等位基因数和有效等位基因数分别为4-17和1-5个,平均每个位点分别为7.9和2.8个。观察杂合度和期望杂合度变化于0.084 0-0.908 2和0.097 9-0.782 6,平均为0.558 7和0.588 8;从位点水平来看,PTest1和PtTX3011存在明显的杂合子缺失,而RPTest11有微小的杂合子缺失,其余4个均未表现出杂合子缺失;从整体水平来看,存在一定杂合子缺失的现象。
2讨论
微卫得位点可实现种间的可转移性,本研究中采用的同属近缘种引物进行筛选,从59对引物中共筛选出引物23对(38.98%),从中筛选出多态性较好的引物7对(11.86%),相比同属马尾松50%的有效扩增而言偏低(蔡娟娟等, 2009),而与油松相比略高(张冬梅等, 2007),这种差异与引物的来源和扩增树种间的亲缘关系远近有关,不同树种之间的亲缘关系远近会对引物的通用性造成影响,因此要尽量合成亲缘关系相对较近的引物(张冬梅等, 2007)。
各引物扩增结果Hardy-Weinberg检测表明,大多数引物显著偏离Hardy-Weinberg检测,这可能是由于零等位基因、有限的样本量和杂合子的缺失造成(Li et al., 2012)。零等位基因即对微卫星位点进行检测时,等位基因带型很弱甚至不能识别,位点与引物结合部位间大片段插入或缺失、DNA质量差、位点突变大幅度改变了产物大小等也能导致零等位基因出现(郭昱嵩等, 2010);其次,本研究是用不同地理起源的群体来分析引物扩增效果,这种不同斑块的样本来源可能也会导致Hardy-Weinberg的偏离,这在西南桦(Betula alnoides)研究中也有报道(Guo et al., 2008);而杂合子的缺失在本研究中只有2个位点表现明显,说明由杂合子的缺失引起的偏离Hardy-Weinberg不显著。虽然存在一定Hardy-Weinberg偏离,但本研究中,各位点间无明显的连锁不平衡,且各位点检测出的等位基因数和有效等位基因数平均达7.9和2.8个,完全可用于进一步的遗传分析研究。此外,本研究所用引物数量较少,筛选出的多态性引物较少,按Koskinen(2004)的研究表明,开展群体研究时,需要增加引物的数量。因此,可进一步通过基因组或EST序列开发SSR标记。
3材料与方法
3.1试验材料
初筛引物模板材料采集于白马河林场云南松母树林种子播种获得的2年生实生苗,复筛引物模板材料来源于云南松不同分布区的5个天然群体,分别为:KM (102.60ºE, 25.07ºN)、XW (104.05ºE, 26.32ºN)、FN (105.33ºE, 23.42ºN)、HQ (100.17ºE, 26.29ºN)、LX (100.15ºE, 23.90ºN)等,先用每个群体各4个个体进行多态性扩增检测,最后5个群体各24个样进行Hardy-Weinberg和多态性分析。
3.2 DNA的提取
云南松(Pinus yunnanensis)基因组总DNA采用植物基因组DNA提取试剂盒(北京Tiangen公司)提取,于-20℃下保存备用。
3.3引物筛选
所用引物来源于同属种共59对,其中47对来源于火炬松(pinus taeda, PT),11对来源于辐射松(pinus radiate, PR),1对来源于北美乔松(pinus strobes, PS) (Elsik et al., 2004; Elsik et al., 2001; Zhou et al., 2002; Kutil and Williams, 2001; Devey et al., 2002; 王鹏良, 2006),引物由上海生物工程有限公司合成,所用试剂包括Taq聚合酶(83.35 mkat/L)、Mg2+ (25.0 mol/L)、dNTP (各2.5 mol/L)、Marker (DL2000)、10×PCR Buffer等均购自TaKaRa公司,其它试剂为国产分析纯。
PCR产物加入6×loading Buffer 2 µL,混匀后取6 µL,以DL100 Marker (TaKaRa公司)为对照,用1.5%琼脂糖凝胶(含EB 0.05 µg/mL),于120 V恒压下电泳40 min (北京六一仪器厂电泳仪)。电泳结束后在Bio Imaging System (GeneGenins公司)上观测并拍照和分析,再3730XL测序列分析仪(美国 ABI公司)自动检测SSR样本,分析Hardy-Weinberg和多态性。
3.4扩增体系及程序
反应体系10 μL,其中模板DNA 30.0 ng,TaqDNA聚合酶 1.0 U,Mg2+ 2.0 mmol/L,dNTPs 0.4 mmol/L,正反引物各 0.2 μmol/L。在下列程序下扩增程:94℃预变性4 min;94℃变性30 s,最适退火温度(依不同引物而异)40 s,72℃延伸45 s,30个循环;72℃延长10 min,4℃保存(张瑞丽等, 2012)。
3.5 数据分析
对自动荧光检测获得的结果,采用GeneMarker1.85软件包提取DNA片段信息,根据ABI GS500 Standard,计算出扩增片段大小,获得各位点的扩增片段。将各位点的扩增进行汇总,利用Convert 1.31 (Glaubitz et 将结果软件转化成Popgene格式,用Popgene 32 (Yeh et al., 1999, http://www.ualberta.ca/~fyeh/popgene.pdf)进行统计分析。
作者贡献
张瑞丽、白青松是本研究的实验设计和实验研究的执行人;许玉兰参与实验设计,数据分析,论文写作与修改;田斌、王大玮参与实验设计,试验结果分析及论文的修改;段安安是项目的构思者及负责人,指导实验设计。全体作者都阅读并同意最终的文本。
致谢
本研究在西南山地森林保育与利用省部共建教育部重点实验室和上海生物工程有限公司完成,由云南省自然科学基金项目(2010CD065)、云南省教育厅重点项目(2010Z042)、国家自然科学基金项目(31260191)、云南省教育厅研究生项目(2011J053)和云南省省院省校教育合作咨询共建重点学科(211015)共同资助。
参考文献
Chen H.Q., Sui C., and Wei J.H., 2009, Summary of strategies for developing SSR primer, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 7(4): 845-851 (陈怀琼, 隋春, 魏建和, 2009, 植物SSR引物开发策略简述, 分子植物育种, 7(4): 845-851)
Cai J.J., and Ji K.S., 2009, Diversity of SSR markers and strategy of genetic linkage map making with masson's pine megagametophyte, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 7(5): 934-940 (蔡娟娟, 季孔庶, 2009, 马尾松大配子体的SSR多样性及其遗传作图策略研究, 分子植物育种, 7(5): 934-940)
Devey M.E., Bell J.C., Uren T.L., and Moran G.F., 2002, A set of microsatellite markers for fingerprinting and breeding application in Pinus radiata, Genome, 45(5): 984-989
http://dx.doi.org/10.1139/g02-064 PMid:12416633
Eckert A.J., Eckert M.L., and Hall B.D., 2010, Effects of historical demography and ecological context on spatial patterns of genetic diversity within Foxtail pine (Pinus balfouriana; Pinaceae) stands located in the Klamath Mountains, California, Am. J. Bot., 97(4): 650-659
http://dx.doi.org/10.3732/ajb.0900099 PMid:21622427
Elsik C.G., Minihan V.T., Hall S.E., Scarpa A.M., and Williams C.G., 2000, Low-copy microsatellite markers for Pinus taeda L., Genome, 43(3): 550-555
PMid:10902720
Elsik C.G., and Williams C.G., 2001, Low-copy microsatellite recovery from a conifer genome. Theor. Appl. Genet.,103(8): 1189-1195
http://dx.doi.org/10.1007/s001220100725
Glaubitz J.C., 2004, Convert: a user-friendly program to reformat diploid genotypic data for commonly used population genetic software packages, Mol. Ecol. Not., 4(2): 309-310
http://dx.doi.org/10.1111/j.1471-8286.2004.00597.x
Guo J.J., Zeng J., Zhou S.L., and Zhao Z.G., 2008, Isolation and characterization of 19 microsatellite markers in a tropical and warm subtropical birch, Betula alnoides Buch.-Ham. ex D. Don, Mol. Ecol. Resour., 8(4): 895-897
http://dx.doi.org/10.1111/j.1755-0998.2008.02101.x PMid:21585922
Guo Y.S., Wang Z.D., Liu C.W., Chen Z.M., and Liu Y., 2010, Isolation and genetic diversity analysis of microsatellites from nine species of familiar Snappers, Redai Haiyang Xuebao (Journal of Tropical Oceanography), 29(3): 82-86 (郭昱嵩, 王中铎, 刘楚吾, 陈志明, 刘筠, 2010, 9种常见笛鲷微卫星位点筛选与遗传多样性分析, 热带海洋学报, 29(3): 82-86)
Kalia R.K., Rai M.K., Kalia S., Singh R., and Dhawan A.K., 2011, Microsatellite markers: an overview of the recent progress in plants, Euphytica, 177(3): 309-334
http://dx.doi.org/10.1007/s10681-010-0286-9
Koskinen M.T., Hirvonen H., Landry P., and Primmer C.R., 2004, The benefits of increasing the number of microsatellites utilized in genetic population studies: An empirical perspective, Hereditas, 141(1): 61-67
http://dx.doi.org/10.1111/j.1601-5223.2004.01804.x PMid:15383073
Krapal A.M., Popa O.P., Iorgu E.I., Costache M., and Popa L.O., 2012, Isolation and characterization of new microsatellite markers for the invasive softshell clam, mya arenaria (L.) (bivalvia: myidae), Int. J. Mol. Sci., 13(2): 2515-2520
http://dx.doi.org/10.3390/ijms13022515 PMid:22408468 PMCid:3292037
Krutovsky K.V., Troggio M., Brown G.R., Jermstad K.D., and Neale D.B., 2004, Comparative mapping in the pinaceae, Genetics, 168(1): 447-461
http://dx.doi.org/10.1534/genetics.104.028381 PMid:15454556 PMCid:1448108
Kutil B.L., and Williams C.G., 2001, Triplet-repeat microsatellites shared among hard and soft pines, J. Hered., 92(4): 327-322
http://dx.doi.org/10.1093/jhered/92.4.327 PMid:11535645
Li R., Yang J., Yang J., and Dao Z.L., 2012, Isolation and characterization of 21 microsatellite loci in cardiocrinum giganteum var. yunnanense (liliaceae), an important economic plant in china, Int. J. Mol. Sci., 13(2):1437-1443
http://dx.doi.org/10.3390/ijms13021437 PMid:22408400 PMCid:3291969
Powell W., Morgante M., Andre C., Hanafey M., Vogel J., Tingey S., and Rafalski A.,1996, The comparison of RFLP, RAPD, AFLP and SSR (microsatellite) markers for germplasm analysis, Mol. Breed., 2: 225-238
http://dx.doi.org/10.1007/BF00564200
Soto A., Robledo-Arnuncio J.J., González-MartíNez S.C., Smouse P.E., and Ali´A.R., 2010, Climatic niche and neutral genetic diversity of the six Iberian pine species: a retrospective and prospective view, Mol. Ecol., 19(7):1396-1409
http://dx.doi.org/10.1111/j.1365-294X.2010.04571.x PMid:20196810
Wang P., 2006, Genetic diversity in three diffcrent year planted offspring stands from masson pine c1onal seed orchard, Thesis for M.S., Nanjing Forestry University, Supervisor: Ji K.S., pp.1-54 (王鹏良, 2006, 马尾松无性系种子园多年份子代遗传多样性分析, 南京林业大学, 导师: 季孔庶, pp.1-54)
You W.Y., Huang H.H., Cheng L.J., Tong Z.K., and Zhu Y.Q., 2010, An SSR molecular labeling technique system for Betula luminifera, Zhejiang Linxueyuan Xuebao (Journal of Zhejiang Forestry College), 27(3): 464-469 (尤卫艳, 黄华宏, 程龙军, 童再康, 朱玉球, 2010, 光皮桦SSR分子标记体系的建立, 浙江林学院学报, 27(3): 464-469)
Zhang D.M., Yang Y., Shen X.H., and Ru G.X., 2007, Selection of primers and establishment of SSR-PCR reaction system on Pinus tabulaeformis Carr, Beijing Linye Daxue Xuebao (Journal of Beijing Forestry University), 29(2) 13-17 (张冬梅, 杨娅, 沈熙环, 茹广欣, 2007, 油松SSR-PCR引物筛选及反应体系的建立, 北京林业大学学报, 29(2): 13-17)
Zhang R.L., Xu Y.L., Wang D.W., Lü X.H., He C.Z., and Duan A.A., 2012, Establishment and optimization of the SSR reaction system for Pinus yunnanensis Using Orthogonal Design, Shengwu Jishu Tongbao (Biotechnology Bulletin), 4: 93-97 (张瑞丽, 许玉兰, 王大玮, 吕学辉, 承忠, 安安, 2012, 云南松SSR-PCR反应体系的建立与优化, 生物技术通报, 4: 93-97)
Zhou Y., Bui T., Auckland L.D., and Williams C.G., 2002, Undermethylated DNA as a source of microsatellites from a conifer genome, Genome, 45(1): 91-99
http://dx.doi.org/10.1139/g01-119 PMid:11908673
.jpg)
.jpg)